guiar a las células por las rutas de diferenciación deseadas

Una guía computacional para guiar a las células por las rutas de diferenciación deseadas
12 de marzo de 2021
Una nueva herramienta de diseño guiada por computadora predice combinaciones de factores de transcripción para una ingeniería más efectiva de tipos de células con uso potencial en terapias celulares y modelado de tejidos.
Por Benjamin Boettner
(BOSTON) – Existe una gran necesidad de generar varios tipos de células para su uso en nuevas terapias para reemplazar los tejidos que se pierden debido a enfermedades o lesiones, o para estudios fuera del cuerpo humano para mejorar nuestra comprensión de cómo funcionan los órganos y tejidos en salud y enfermedad. Muchos de estos esfuerzos comienzan con células madre pluripotentes inducidas por humanos (iPSC) que, en teoría, tienen la capacidad de diferenciarse en prácticamente cualquier tipo de célula en las condiciones de cultivo adecuadas. El Premio Nobel de 2012 otorgado a Shinya Yamanaka reconoció su descubrimiento de una estrategia que puede reprogramar células adultas para que se conviertan en iPSC proporcionándoles un conjunto definido de factores de transcripción reguladores de genes (TF). Sin embargo,
Si bien la expresión de TF específicos de tipo celular en iPSC es la tecnología de conversión celular más utilizada, la eficiencia de guiar iPSC a través de diferentes “etapas de linaje” hasta el estado diferenciado completamente funcional de, por ejemplo, un corazón, cerebro o sistema inmunológico actualmente son bajas, principalmente porque las combinaciones de TF más efectivas no se pueden identificar fácilmente. Los TF que indican a las células que pasen por un proceso de diferenciación celular específico se unen a regiones reguladoras de genes para controlar su expresión en el genoma. Sin embargo, múltiples TF deben funcionar en el contexto de redes reguladoras de genes (GRN) más grandes para impulsar la progresión de las células a través de sus linajes hasta que se alcance el estado diferenciado final.
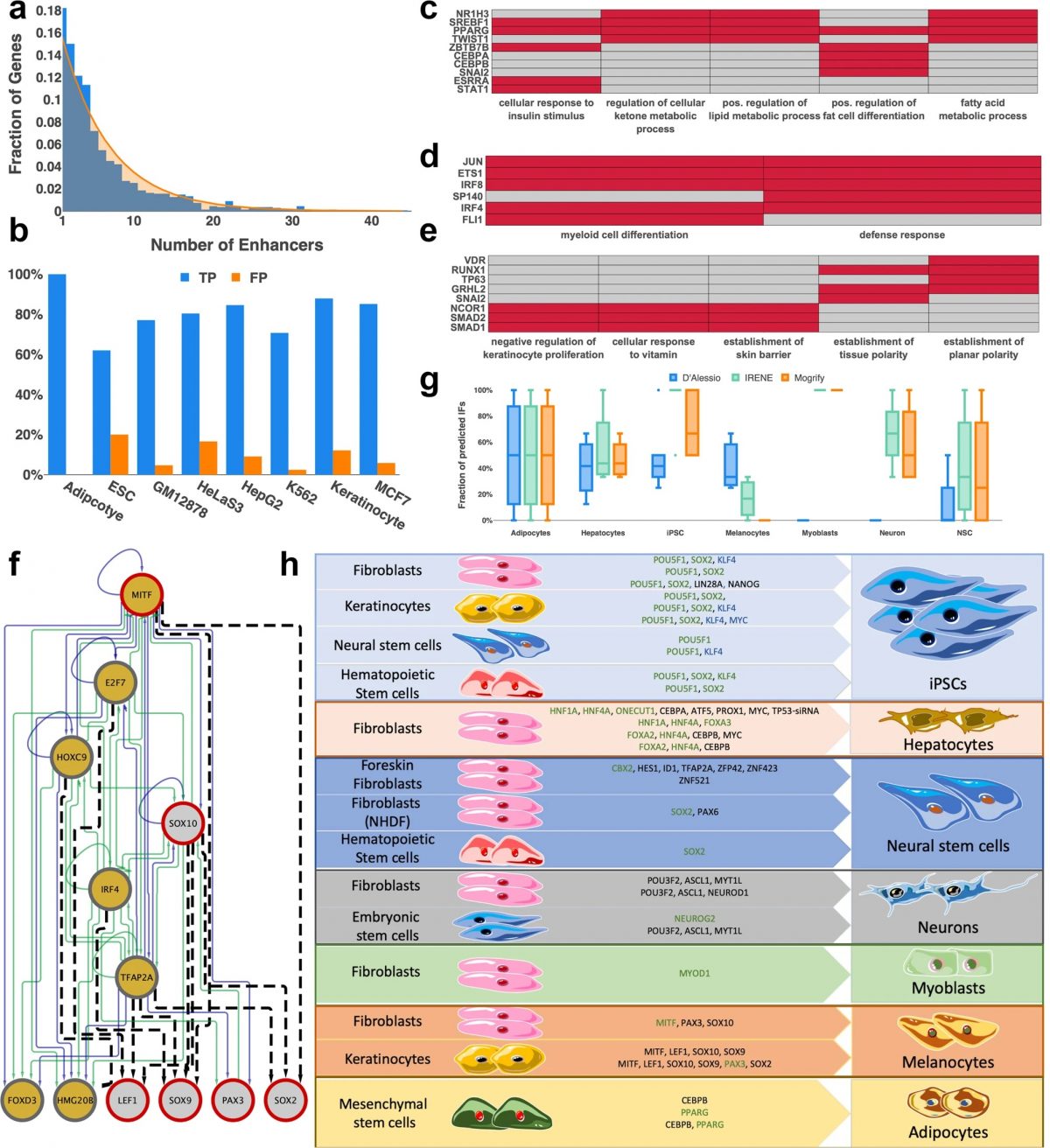
a El número de potenciadores por gen (azul) en todas las redes sigue una distribución exponencial (naranja). b Evaluación comparativa de redes reconstruidas frente a datos TF ChIP-Seq específicos del tipo de célula para 8 tipos de células / líneas de células. Los verdaderos positivos (TP, azul) representan las interacciones que están presentes en los GRN reconstruidos y se validan experimentalmente mediante datos TF ChIP-Seq específicos del tipo de célula. Las interacciones validadas por los datos de TF ChIP-Seq solo perfilados en tipos de células distintos del que se está considerando se consideran falsos positivos (FP, naranja). n = 13 (adipocito), 69 (ESC), 236 (GM12878), 238 (HeLaS3), 292 (HepG2), 53 (K562), 37 (queratinocito) y 178 (MCF7) interacciones. c – eLa mayoría-altamente enriquecido de genes significativa ontología términos de las redes reconstruidas de c adipocitos, d células asesinas naturales y e las células epiteliales mamarias. Para los adipocitos y las células epiteliales mamarias, se representan los 5 términos GO superiores. Para las células asesinas naturales, solo dos términos se enriquecieron significativamente. Las celdas correspondientes a los TF que son y no son relevantes para un término de GO particular están coloreadas en rojo y gris, respectivamente. FSubred de melanocitos reconstruida que incluye todos los IF validados experimentalmente (borde rojo) y predichos (oro) para la conversión de fibroblastos en melanocitos. La regulación del potenciador y del promotor (verde) se distingue de la regulación solo del potenciador (azul). Las interacciones pronosticadas de las matrices de peso de posición utilizando Homer se representan como líneas discontinuas negras. gramoRecuperación de IF validados experimentalmente en siete tipos de células diana utilizando IRENE (verde), Mogrify (naranja) y el método de d’Alessio et al. (azul). La fracción de IF recuperados en múltiples combinaciones de conversiones celulares se representa como diagramas de caja. La mediana está representada por una línea continua dentro de los cuadros. Los límites inferior y superior de los cuadros son el primer y tercer cuartil, respectivamente. Los bigotes se extienden hasta 1,5 veces el rango intercuartílico o el valor mínimo / máximo. Los puntos corresponden a valores atípicos. n = 3 (adipocitos), 4 (hepatocitos), 10 (iPSC), 3 (melanocitos), 1 (mioblastos), 4 (neurona) y 4 (NSC) combinaciones de factores de conversión celular. hEnriquecimiento de factores instructivos predichos en combinaciones de FI validadas experimentalmente. Los FI pronosticados se resaltan en verde, mientras que los TF que fueron reemplazados por otro FI validado y más eficiente están resaltados en azul. Los TF no predichos por IRENE están coloreados en negro.

George-Church-
Ahora, un esfuerzo de colaboración dirigido por George Church , Ph.D. en el Instituto Wyss de Harvard para Ingeniería de Inspiración Biológica y en la Escuela de Medicina de Harvard (HMS), y Antonio del Sol, Ph.D., quien dirige los grupos de Biología Computacional en CIC bioGUNE, miembro de la Alianza Vasca de Investigación y Tecnología, en España, y en el Centro de Biomedicina de Sistemas de Luxemburgo (LCSB, Universidad de Luxemburgo), ha desarrollado un herramienta de diseño llamada IRENE, que ayuda significativamente a aumentar la eficiencia de las conversiones de células al predecir combinaciones altamente efectivas de TF específicos del tipo de célula. Al combinar IRENE con un sistema de integración genómica que permite la expresión robusta de TF seleccionados en iPSC, el enfoque del equipo generó un mayor número de células asesinas naturales (utilizadas en terapias inmunes) y melanocitos (utilizados en injertos de piel) que otros métodos. Por primera vez desde el punto de vista científico, también generó células epiteliales mamarias mamarias, cuya disponibilidad es muy deseable para la repoblación del tejido mamario extirpado quirúrgicamente.Comunicaciones de la naturaleza .

Antonio del Sol Mesa
“En nuestro grupo, el estudio se basó naturalmente en el proyecto ‘TFome’ , que reunió una biblioteca completa que contiene 1564 TF humanos como un recurso poderoso para la identificación de combinaciones de TF con capacidades mejoradas para reprogramar iPSC humanas a diferentes tipos de células diana”, dijo Iglesia miembro de Wyss Core Faculty. “La eficacia de este algoritmo computacional impulsará varios de nuestros esfuerzos de ingeniería de tejidos en el Instituto Wyss y HMS, y como recurso abierto puede hacer lo mismo para muchos investigadores en este campo floreciente”. Church es el líder de la plataforma de Biología Sintética del Instituto Wyss y profesor de Genética en HMS y de Ciencias y Tecnología de la Salud en Harvard y MIT.
La eficacia de este algoritmo computacional impulsará varios de nuestros esfuerzos de ingeniería de tejidos en el Instituto Wyss y HMS, y como recurso abierto puede hacer lo mismo para muchos investigadores en este campo floreciente.
Herramientas arriba guiar a las células por las rutas de diferenciación deseadas
Se han desarrollado varias herramientas computacionales para predecir combinaciones de TF para conversiones celulares específicas, pero casi exclusivamente se basan en el análisis de patrones de expresión génica en muchos tipos de células. En estos enfoques faltaba una visión del paisaje epigenético, la organización del genoma en sí mismo alrededor de los genes y en la escala de secciones cromosómicas completas que va mucho más allá de la secuencia del ADN genómico desnudo.
“El panorama epigenético cambiante en la diferenciación de células predice áreas en el genoma que experimentan cambios físicos que son críticos para que los TF clave obtengan acceso a sus genes diana. El análisis de estos cambios puede informar con mayor precisión sobre los GRN y sus TF participantes que impulsan conversiones celulares específicas ”, dijo el coautor Evan Appleton, Ph.D. Appleton es un becario postdoctoral en el grupo de Church que unió fuerzas con Sascha Jung, Ph.D., del grupo de del Sol en el nuevo estudio. “Nuestros colaboradores en España habían desarrollado un enfoque computacional que integraba esos cambios epigenéticos con cambios en la expresión génica para producir combinaciones críticas de TF como resultado, que estábamos en una posición ideal para probar”.
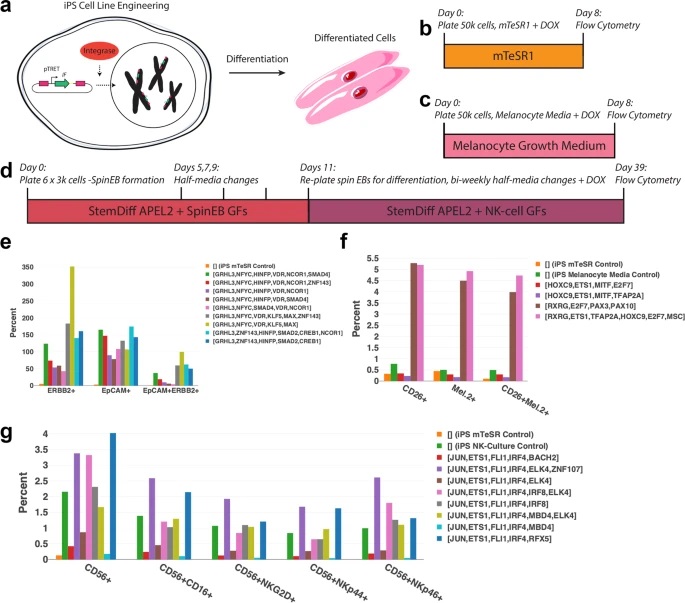
a Se crearon líneas de iPS estables para todos los experimentos de diferenciación antes de la diferenciación mediante la integración genómica para asegurar una expresión alta y estable de IF utilizando el TFome humano. b Protocolo para diferenciar las células epiteliales mamarias humanas (HMEC) de las células iPS humanas (hiPSC). c Protocolo para diferenciar melanocitos de hiPSC. d Protocolo para diferenciar las células NK de las hiPSC. (GF: factor de crecimiento, DOX: doxiciclina). e – g Eficiencia de diferenciación de ( e ). HMEC, melanocitos f y gCélulas NK de hiPSC para diversas combinaciones de IF generadas a partir de IRENE. La eficiencia se define como el número de células con marcador positivo / doble positivo dividido por el número de células en placa. n = 1 experimento de tres muestras agrupadas biológicamente independientes.
El equipo utilizó su enfoque computacional “Modelo de red reguladora de genes integradores” (IRENE) para reconstruir las iPSC que controlan GRN, y luego se centró en tres tipos de células diana con relevancia clínica para validar experimentalmente las combinaciones de TF priorizadas por IRENE. Para entregar combinaciones de TF en iPSC, implementaron un sistema de integración genómica basado en transposones que puede integrar múltiples copias de un gen que codifica un TF en el genoma, lo que permite que todos los factores de una combinación se expresen de manera estable. Los transposones son elementos de ADN que pueden saltar de una posición del genoma a otra o, en este caso, de un fragmento de ADN proporcionado de forma exógena al genoma. guiar a las células por las rutas de diferenciación deseadas guiar a las células por las rutas de diferenciación deseadas guiar a las células por las rutas de diferenciación deseadas
“Nuestro equipo de investigación, compuesto por científicos de LCSB y CIC bioGUNE, tiene una dilatada experiencia en el desarrollo de métodos computacionales para facilitar la conversión celular. IRENE es un recurso adicional en nuestra caja de herramientas y uno para el cual la validación experimental ha demostrado que aumenta sustancialmente la eficiencia en la mayoría de los casos probados ”, autor correspondiente Del Sol, quien es profesor en LCSB y CIC bioGUNE. “Nuestra investigación fundamental debería beneficiar en última instancia a los pacientes, y estamos encantados de que IRENE pueda mejorar la producción de fuentes celulares fácilmente utilizables en aplicaciones terapéuticas, como el trasplante celular y las terapias génicas”. guiar a las células por las rutas de diferenciación deseadas guiar a las células por las rutas de diferenciación deseadas
Validación de la herramienta de diseño guiado por computadora en celdas
Los investigadores eligieron las células epiteliales mamarias humanas (HMEC) como primer tipo de célula. Hasta ahora, las HMEC se obtienen de un entorno de tejido, se disocian y se trasplantan a uno en el que se ha resecado tejido mamario. Las HMEC generadas a partir de las células de los pacientes, a través de una etapa intermedia de iPSC, podrían proporcionar un medio para una regeneración del tejido mamario menos invasiva y más eficaz. Una de las combinaciones que generó IRENE permitió al equipo convertir el 14% de las iPSC en HMEC diferenciadas en medios de cultivo específicos de iPSC, lo que demuestra que los TF proporcionados fueron suficientes para impulsar la conversión sin la ayuda de factores adicionales.
El panorama epigenético cambiante en la diferenciación de células predice áreas en el genoma que experimentan cambios físicos que son críticos para que los factores de transcripción clave obtengan acceso a sus genes diana. El análisis de estos cambios puede informar con mayor precisión sobre las redes reguladoras de genes y sus factores de transcripción participantes que impulsan conversiones celulares específicas.
Luego, el equipo centró su atención en los melanocitos, que pueden proporcionar una fuente de células en los injertos celulares para reemplazar la piel dañada. Esta vez realizaron la conversión celular en medio de destino de melanocitos para mostrar que los TF seleccionados funcionan en condiciones de cultivo optimizadas para el tipo de célula deseado. Dos de cada cuatro combinaciones pudieron aumentar la eficiencia de la conversión de melanocitos en un 900% en comparación con las iPSC cultivadas en el medio de destino sin los TF. Finalmente, los investigadores compararon combinaciones de TF priorizadas por IRENE para generar células asesinas naturales (NK) con un método de diferenciación de vanguardia basado únicamente en las condiciones del cultivo celular. Se ha descubierto que las células NK inmunes mejoran el tratamiento de la leucemia.
“Este novedoso enfoque computacional podría facilitar en gran medida una variedad de esfuerzos de ingeniería de células y tejidos en el Instituto Wyss y en muchos otros sitios alrededor del mundo. Este avance debería ampliar enormemente nuestra caja de herramientas a medida que nos esforzamos por desarrollar nuevos enfoques en la medicina regenerativa para mejorar la vida de los pacientes ”, dijo el director fundador de Wyss, Donald Ingber , MD, Ph.D., quien también es profesor de Biología Vascular Judah Folkman en HMS. y Boston Children’s Hospital, y profesor de bioingeniería en la Escuela de Ingeniería y Ciencias Aplicadas John A. Paulson de Harvard.
El estudio también fue escrito por el bioinformático Muhammad Ali, Ph.D., de la Universidad de Luxemburgo, y financiado por el programa de investigación e innovación Horizonte 2020 de la Unión Europea con la subvención # 643417, el programa FunGCAT de la Oficina del Director de Inteligencia Nacional. Actividad de Proyectos de Investigación Avanzada, a través de la Oficina de Investigación del Ejército, bajo el premio # W911NF-17-2-0089 y la Fundación Caritativa EGL. Church es cofundador y participa en GC Therapeutics, que comercializa el proyecto TFome.


