NIH, La IA predice con precisión el plegamiento de proteínas

La inteligencia artificial predice con precisión el plegamiento de proteínas
Publicado en por el Dr. Francis Collins
RoseTTAfold
Las proteínas son los caballos de batalla de la célula. Mapear las formas precisas de los más importantes de estos caballos de batalla ayuda a desbloquear sus funciones de soporte vital o, en el caso de una enfermedad, el potencial de disfunción. Si bien la secuencia de aminoácidos de una proteína proporciona la base para su estructura 3D, deducir el mapa átomo por átomo de los principios de la mecánica cuántica ha estado más allá de la capacidad de los programas de computadora, hasta ahora.
En un estudio reciente en la revista Science , los investigadores informaron que han desarrollado enfoques de inteligencia artificial para predecir la estructura tridimensional de las proteínas en un tiempo récord, basándose únicamente en sus secuencias de aminoácidos unidimensionales [1]. Este enfoque innovador no solo ayudará a los investigadores en el laboratorio, sino que también guiará a los desarrolladores de medicamentos a encontrar formas más seguras y efectivas de tratar y prevenir enfermedades.
Este nuevo avance apoyado por los NIH ahora está disponible gratuitamente para científicos de todo el mundo. De hecho, ya ha ayudado a resolver estructuras de proteínas especialmente desafiantes en casos en los que faltaban datos experimentales y otros métodos de modelado no habían sido suficientes para obtener una respuesta final. Ahora también puede proporcionar información estructural clave sobre proteínas para las que aún no se dispone de datos de imágenes más costosos y que consumen más tiempo.

Minkyung Baek,
El nuevo trabajo proviene de un grupo dirigido por David Baker y Minkyung Baek, Instituto de Diseño de Proteínas de la Universidad de Washington, Seattle. Durante el transcurso de la pandemia, el equipo de Baker ha estado trabajando arduamente para diseñar terapias prometedoras para COVID-19. También han estado trabajando para diseñar proteínas que podrían ofrecer nuevas y prometedoras formas de tratar el cáncer y otras afecciones. Como parte de este esfuerzo, han desarrollado nuevos enfoques computacionales para determinar con precisión cómo una cadena de aminoácidos, que son los componentes básicos de las proteínas, se plegará en el espacio para formar una proteína terminada.

David Baker in lab social
Pero la capacidad de predecir la estructura o forma precisa de una proteína a partir de su secuencia solo había demostrado ser un problema difícil de resolver a pesar de décadas de esfuerzo. En busca de una solución, equipos de investigación de todo el mundo se han reunido cada dos años desde 1994 en las reuniones de Evaluación crítica de predicción de estructuras (CASP). En estas reuniones, los equipos compiten entre sí con el objetivo de desarrollar métodos computacionales y software capaz de predecir cualquiera de los 200 millones o más de estructuras proteicas de la naturaleza a partir de secuencias con la mayor precisión.
El año pasado, una empresa con sede en Londres llamada DeepMind revolucionó el mundo de la biología estructural con su entrada en CASP llamada AlphaFold. (AlphaFold fue uno de los avances científicos del año en 2020 ). Demostraron que su enfoque de inteligencia artificial, que aprovechó las 170.000 proteínas con estructuras conocidas en un proceso reiterativo llamado aprendizaje profundo, podía predecir la estructura de las proteínas con una precisión asombrosa. De hecho, podría predecir la mayoría de las estructuras de proteínas casi con tanta precisión como otras técnicas de mapeo de proteínas de alta resolución, incluidas las estrategias actuales de cristalografía de rayos X y crio-EM .
El rendimiento de DeepMind mostró lo que era posible, pero debido a que los avances fueron realizados por una empresa de aprendizaje profundo líder en el mundo, los detalles sobre cómo funcionaba no se pusieron a disposición del público en ese momento. Los hallazgos dejaron a Baker, Baek y otros ansiosos por aprender más y ver si podían replicar la impresionante capacidad predictiva de AlphaFold fuera de una empresa con tan buenos recursos.
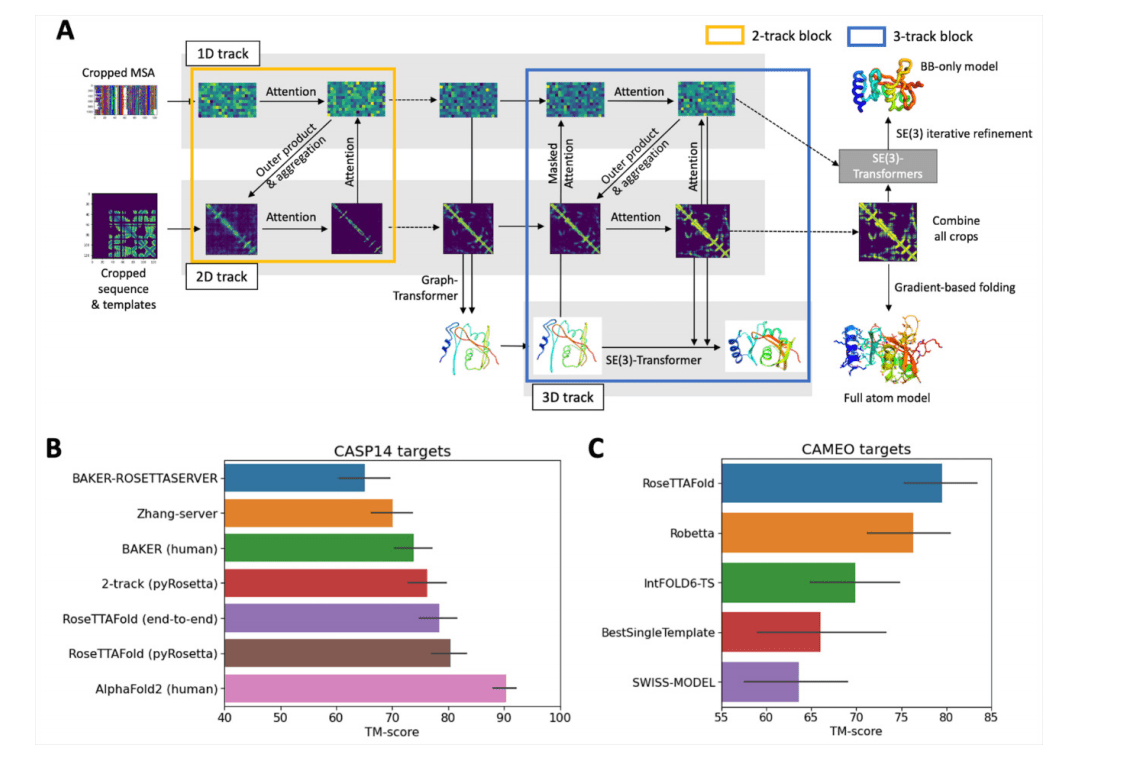
En el nuevo trabajo, el equipo de Baker y Baek ha logrado un progreso asombroso, utilizando solo una fracción de la potencia de procesamiento computacional y el tiempo que requiere AlphaFold. El nuevo software, llamado RoseTTAFold, también se basa en un enfoque de aprendizaje profundo. En el aprendizaje profundo, las computadoras buscan patrones en grandes colecciones de datos. A medida que comienzan a reconocer relaciones complejas, algunas conexiones en la red se fortalecen mientras que otras se debilitan. La red terminada se compone típicamente de múltiples capas de procesamiento de información, que operan sobre los datos para devolver un resultado, en este caso, una estructura de proteína.
Dada la complejidad del problema, en lugar de utilizar una única red neuronal, RoseTTAFold se basa en tres. La red neuronal de tres pistas integra y procesa simultáneamente información unidimensional de la secuencia de proteínas, información bidimensional sobre la distancia entre los aminoácidos y la estructura atómica tridimensional, todo a la vez. La información de estas pistas separadas fluye hacia adelante y hacia atrás para generar modelos precisos de proteínas rápidamente a partir de la información de la secuencia sola, incluidas las estructuras en complejo con otras proteínas.
Tan pronto como los investigadores tuvieron lo que pensaban que era un enfoque de trabajo razonable para resolver las estructuras de las proteínas, comenzaron a compartirlo con sus colegas biólogos estructurales. En muchos casos, quedó claro de inmediato que RoseTTAFold funcionaba muy bien. Es más, se ha puesto a trabajar para resolver desafiantes problemas de biología estructural que habían molestado a los científicos durante muchos años con métodos anteriores.
RoseTTAFold ya ha resuelto cientos de nuevas estructuras de proteínas, muchas de las cuales representan proteínas humanas poco conocidas. La representación 3D de un complejo que muestra una proteína humana llamada interleucina-12 en un complejo con su receptor (imagen de arriba) es solo un ejemplo. Los investigadores han generado otras estructuras directamente relevantes para la salud humana, incluidas algunas relacionadas con el metabolismo de los lípidos, las afecciones inflamatorias y el cáncer. El programa ya está disponible en la web y ha sido descargado por docenas de equipos de investigación de todo el mundo. NIH, La IA predice con precisión el plegamiento de proteínas
Cryo-EM y otros métodos de mapeo experimentales seguirán siendo esenciales para resolver las estructuras de proteínas en el laboratorio. Pero con los avances en inteligencia artificial demostrados por RoseTTAFold y AlphaFold, que ahora también se lanzó en una versión de código abierto y se informó en la revista Nature [2], los investigadores ahora pueden hacer predicciones de la estructura de proteínas críticas en sus escritorios. Esta nueva habilidad será de gran ayuda para los estudios de ciencias básicas y tiene un gran potencial para acelerar los avances terapéuticos que salvan vidas.
Referencias :
[1] Predicción precisa de las estructuras e interacciones de las proteínas mediante una red neuronal de tres pistas . Baek M, DiMaio F, Anishchenko I, Dauparas J, Grishin NV, Adams PD, Read RJ, Baker D., et al. Ciencia. 15 de julio de 2021: eabj8754.
[2] Predicción de la estructura de proteínas de alta precisión con AlphaFold . Jumper J, Evans R, Pritzel A, Green T, Senior AW, Kavukcuoglu K, Kohli P, Hassabis D. et al. Naturaleza. 2021 15 de julio.
Tintas L : NIH, La IA predice con precisión el plegamiento de proteínas
Biología estructural (Instituto Nacional de Ciencias Médicas Generales / NIH)
Las estructuras de la vida (NIGMS)
Baker Lab (Universidad de Washington, Seattle)
CASP 14 (Universidad de California, Davis)
Apoyo de los NIH: Instituto Nacional de Alergias y Enfermedades Infecciosas; Instituto Nacional de Ciencias Médicas Generales