Detecta el cáncer hasta cuatro años antes que los métodos convencionales
La detección temprana tiene el potencial de reducir la mortalidad por cáncer
PanSeer detecta el cáncer hasta cuatro años antes del diagnóstico

Un-análisis-de-sangre-podría-detectar-los-tipos-de-cáncer-más-comunes-cuatro-años-antes-que-los-métodos-actuales
La detección temprana tiene el potencial de reducir la mortalidad por cáncer, pero una prueba de detección efectiva debe demostrar la detección de cáncer asintomático años antes del diagnóstico convencional en un estudio longitudinal. En el Estudio Longitudinal de Taizhou (TZL), 123,115 sujetos sanos proporcionaron muestras de plasma para el almacenamiento a largo plazo y luego fueron monitoreados para detectar la aparición de cáncer. Aquí presentamos los resultados preliminares de PanSeer, un análisis de sangre no invasivo basado en la metilación del ADN del tumor circulante, en muestras de plasma TZL de 605 individuos asintomáticos, 191 de los cuales fueron diagnosticados más tarde con cáncer de estómago, esófago, colorrectal, pulmón o hígado dentro de los cuatro años de extracción de sangre. También analizamos muestras de plasma de 223 pacientes con cáncer adicionales, más 200 tumores primarios y tejidos normales. Mostramos que PanSeer detecta cinco tipos comunes de cáncer en el 88% (IC del 95%: 80-93%) de pacientes post diagnóstico con una especificidad de 96% (IC 95%: 93-98%), también demostramos que PanSeer detecta cáncer en 95% (IC 95%: 89-98%) de individuos asintomáticos quienes fueron diagnosticados posteriormente, aunque se requieren futuros estudios longitudinales para confirmar este resultado. Estos resultados demuestran que el cáncer puede detectarse de manera no invasiva hasta cuatro años antes del estándar de atención actual.
Detecta el cáncer hasta cuatro años antes que los métodos convencionales
Detección temprana no invasiva de cáncer cuatro años antes del diagnóstico convencional mediante un análisis de sangre.
Los cánceres en etapa tardía a menudo carecen de una opción de tratamiento eficaz 1 , 2 . Las tasas de supervivencia aumentan significativamente cuando el cáncer se identifica en las primeras etapas, ya que el tumor se puede extirpar quirúrgicamente o tratar con regímenes farmacológicos más leves 3 ; la supervivencia promedio de 5 años en la etapa inicial es del 91%, mientras que la supervivencia promedio de 5 años en la etapa tardía es del 26% 4 . Por lo tanto, la detección de tumores en la etapa más temprana posible es de suma importancia para el tratamiento del cáncer. Actualmente, existe un número limitado de pruebas de detección para algunos tipos de cáncer, incluida la colonoscopia 5 , el antígeno prostático específico 6 , la mamografía 7 y la citología cervical 8. Sin embargo, la eficacia de algunas pruebas ha sido cuestionada 9 , y muchos pacientes no siguen las pautas médicas para la detección 10 . La mayoría de los tipos de cáncer actualmente carecen de una opción efectiva de detección temprana no invasiva 11 . Es importante destacar que una demostración formal de detección temprana requiere recolectar muestras años antes del diagnóstico convencional de cáncer, lo cual solo es factible con el seguimiento longitudinal de un gran número de individuos sanos e identificar la fracción muy pequeña que desarrolla cáncer con el tiempo (a la tasa de incidencia de cáncer en la población en general).
Recientemente, el ADN tumoral circulante (ADNc) en el plasma sanguíneo se ha convertido en un prometedor biomarcador de cáncer 12 . Se ha demostrado que el ADNc tiene utilidad para la detección no invasiva del cáncer 13 , 14 , 15 , 16 , 17 , el tratamiento personalizado del cáncer en etapa tardía 18 y el monitoreo residual del cáncer durante y después del tratamiento 19 , 20 . Sin embargo, los estudios de detección actuales se han centrado principalmente en detectar el cáncer en pacientes que ya han sido diagnosticados 21 , aunque algunos estudios han demostrado la detección del cáncer antes del diagnóstico convencional en tipos de cáncer limitados 13 , 14., 17 . Si bien el ADNc tiene el potencial para un diagnóstico temprano, varias limitaciones dificultan esta tarea. La cantidad de ADN del cáncer en plasma es limitada, especialmente en las primeras etapas; Esto podría limitar la sensibilidad 22 . Los métodos de detección de mutaciones de ADNc típicos pueden ser propensos a errores, lo que lleva a una especificidad reducida; La naturaleza evolutiva del cáncer también se traduce en una cantidad exorbitante de posibles mutaciones que se deben evaluar para lograr un biomarcador constante 23 . Si bien el uso de 5-metilcitosina como biomarcador puede abordar algunas de estas preocupaciones debido a su mayor consistencia en las muestras de cáncer 24 , el proceso de conversión de bisulfito utilizado para interrogar la metilación del ADN daña el ADN 25. Un método de interrogación con una alta tasa de conversión molecular y un conjunto consistente de biomarcadores de cáncer es esencial para garantizar una alta sensibilidad 26 .
Aquí, describimos PanSeer, una prueba de detección de cáncer a base de sangre, y demostramos la detección temprana del cáncer utilizando un conjunto único de muestras recolectadas como parte del Estudio Longitudinal Taizhou (TZL) 27 . PanSeer interroga las firmas de metilación específicas del cáncer y demuestra la detección temprana de múltiples tipos de cáncer hasta cuatro años antes del diagnóstico convencional en un estudio longitudinal retrospectivo a gran escala.
Resultados
Detecta el cáncer hasta cuatro años antes que los métodos convencionales
Desarrollo de ensayos PanSeer
Definimos un conjunto de sitios CpG metilados de manera diferencial utilizando microarrays disponibles públicamente y datos de secuenciación de bisulfito de genoma completo (WGBS) de The Cancer Genome Atlas (TCGA) 28 y regiones genómicas que se sabe que están relacionadas con el cáncer en la literatura 29 , 30 , 31 , 32 , 33 , 34 , 35 , así como datos internos de secuenciación reducida de bisulfito de representación reducida (RRBS) de una variedad de tejidos cancerosos. A partir de estas fuentes, compilamos un panel específico de 595 regiones genómicas (Datos suplementarios 1 ) para su posterior interrogatorio en muestras de plasma.
Intentamos interrogar a estos objetivos en un solo ensayo con alta precisión y eficiencia. El ADN tumoral tiende a ser raro en el plasma, especialmente en pacientes con cáncer en estadio temprano; Debido a que los métodos convencionales para secuenciar la construcción de bibliotecas que incorporan conversión de bisulfito y ligadura bicatenaria típicamente tienen una alta tasa de pérdida de ADN 36 , la sensibilidad de detección puede ser limitada. Por lo tanto, elegimos un método de construcción de la biblioteca Singlera utilizando PCR semi-dirigida. La PCR semi-dirigida requiere solo un único evento de ligadura 37 , 38 , 39 y un solo cebador de PCR por amplicón 40, permitiendo el recuento de una sola molécula a una tasa de recuperación molecular más alta que los métodos convencionales; Esto le da al ensayo PanSeer el potencial de lograr una alta sensibilidad incluso en los cánceres en etapa temprana.
Los métodos de detección basados en metilación previos generalmente se han dirigido a un pequeño número de regiones a gran profundidad a través de PCR 41 , o a un gran número de regiones a baja profundidad a través de la secuenciación de bisulfito del genoma completo (WGBS) o RRBS 42 . Más recientemente, se han descrito técnicas para apuntar a un gran número de regiones con mayor profundidad de secuenciación 43 , 44 , 45. El ensayo PanSeer interroga 595 regiones a gran profundidad; Esto reduce los efectos de la variabilidad del paciente o la deserción del objetivo. Para demostrar la solidez del ensayo PanSeer, realizamos estudios de límite de detección agregando ADN de línea celular de cáncer fragmentado (HT-29) en muestras de plasma sanas agrupadas. Demostramos que el ensayo PanSeer puede detectar picos hasta una fracción de ADN del cáncer de 0.1% (consulte la sección “Métodos”, Fig. Complementaria 1 ); Como se demostró en estudios previos, se puede lograr un alto poder de detección del cáncer con un enfoque de modelado combinatorio 19 .
Identificación y anotación de marcador PanSeer
Con el fin de identificar aún más un conjunto de objetivos genómicos informativos que podrían diferenciar el tejido canceroso del tejido sano, adquirimos un conjunto de 200 muestras de ADN aisladas de cáncer fresco y tejido sano de BioChain, un proveedor comercial de muestras biológicas. Procesamos estas muestras usando el ensayo PanSeer e identificamos un conjunto de 477 regiones metiladas diferencialmente (DMR, ver sección “Métodos”). Para garantizar que las señales identificadas se originaran en el tejido canceroso, limitamos todos los análisis posteriores de muestras de ADN sin células procesadas por el ensayo PanSeer a estas 477 DMR específicas para el cáncer (Datos complementarios 1 , ver sección “Métodos”); estas regiones se asociaron con 657 genes y 10,613 sitios CpG.
Luego buscamos anotar las regiones genómicas presentes en el ensayo PanSeer que podrían discriminar entre tejido sano y tejido canceroso. Como era de esperar, el clasificador PanSeer utilizó muchos genes o familias de genes conocidos relacionados con el cáncer, incluidos los genes de la familia FOX 46 , los genes de la familia HOX 47 , los genes de la familia NKL 48 , los genes de la familia PAX 49 y los genes de la familia TBX 50 . Algunos genes se han utilizado previamente para el diagnóstico de cáncer no invasivo en plasma, como SEPT9 y SHOX2 25 , 51 . Análisis de los términos GO 52Los genes PanSeer mostraron que muchos genes estaban asociados con la actividad de unión al ADN o factor de transcripción, lo que implica que el estado de metilación de estos genes puede contribuir a los cambios en la expresión génica asociados con el cáncer. En línea con las expectativas basadas en el diseño del panel, no observamos ninguna diferencia importante en la representación de genes en diferentes tipos de tejidos cancerosos, ya que los objetivos presentes en el ensayo PanSeer se habían seleccionado previamente para representar un conjunto de genes de cáncer metilados de forma aberrante ubicuamente.
Diseño del estudio Detecta el cáncer hasta cuatro años antes que los métodos convencionales
Detecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales
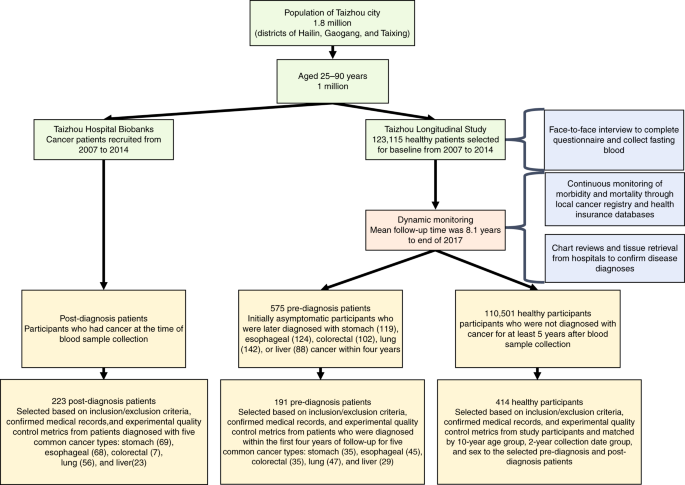
Como parte del estudio TZL 27 , 123,115 sujetos sanos de 25 a 90 años proporcion aron muestras de sangre para el almacenamiento a largo plazo de 2007 a 2014; Estos individuos fueron monitoreados indefinidamente para detectar la aparición de cáncer a través de enlaces con registros locales de cáncer y bases de datos de seguros de salud. A fines de 2017, un total de 575 sujetos inicialmente sanos (que se presentaron como asintomáticos) fueron diagnosticados con uno de los cinco tipos de cáncer comunes (estómago, esófago, colorrectal, pulmón o hígado) dentro de los 4 años posteriores a la extracción de sangre inicial (Fig. 1 ) Se eligieron estos cinco tipos de cáncer porque tenían altas tasas de incidencia en la cohorte de Taizhou y, en combinación, representan la mortalidad más alta en China 53. Al interrogar retrospectivamente las muestras de sangre recolectadas inicialmente, pudimos evaluar si se podía identificar el cáncer antes de los métodos de diagnóstico convencionales.
El diagrama de flujo muestra el reclutamiento, la encuesta de referencia, la recolección de muestras y el seguimiento de cohortes para TZL. Se seleccionaron pacientes calificados antes del diagnóstico y participantes sanos de la cohorte TZL y se seleccionaron pacientes calificados después del diagnóstico de los biobancos del hospital local de Taizhou; Se procesaron 328 muestras, pero luego se excluyeron por no cumplir con los criterios de inclusión o por fallar las métricas de control de calidad.
Primero seleccionamos 221 muestras de pre-diagnóstico de 575 pacientes inicialmente asintomáticos que luego fueron diagnosticados con cáncer de estómago, esófago, colorrectal, pulmón o hígado dentro de los 4 años que cumplían con los criterios de inclusión, y 221 muestras sanas de 110,501 participantes sanos no diagnosticados con cáncer durante al menos 5 años, emparejado individualmente por grupo de edad de 10 años con distribución de edad similar (Figura 2 complementaria ), sexo y fecha de recolección según los criterios de inclusión (consulte la sección “Métodos”, Información complementaria Tabla 1 ). Luego, recolectamos 357 muestras posteriores al diagnóstico y 357 muestras sanas combinadas individualmente por grupo de edad de 10 años, sexo y fecha de recolección de las muestras posteriores al diagnóstico según los criterios de inclusión (consulte la sección “Métodos”, Tabla complementaria 1) Se eliminaron muestras con un número bajo de moléculas de ADN de mapeo único (así como su muestra sana o cancerosa emparejada), dejando 191 muestras pre-diagnóstico, 223 muestras post-diagnóstico y 414 muestras sanas (ver sección “Métodos”). La mediana de edad de los pacientes fue de 62 años (de 35 a 85 años) (Datos complementarios 2 ). Las muestras se dividieron aleatoriamente en un conjunto de entrenamiento para el desarrollo del modelo de conjunto y un conjunto de prueba de exclusión independiente para la validación del modelo en una proporción igual usando un generador de números aleatorios. Las muestras del conjunto de prueba se reservaron hasta que se completó el desarrollo del modelo y los parámetros del modelo se bloquearon utilizando solo las muestras del conjunto de entrenamiento. DDetecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales
etecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales
Desarrollo de un clasificador de regresión logística.
Detecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales
El ensayo PanSeer interroga a 11,787 sitios CpG en 595 regiones del genoma 34 utilizando una mediana de 12 ng de ADN plasmático, aproximadamente 2 millones de lecturas de secuencia y un mínimo de 200,000 moléculas de ADN únicas mapeadas. Primero calculamos la fracción de metilación promedio (AMF) en cada región genómica objetivo para cada muestra (ver sección “Métodos”). Luego desarrollamos un método de aprendizaje automático para clasificar las muestras como derivadas de pacientes sanos o pacientes con cáncer (consulte la sección “Métodos”). Utilizamos las 207 muestras sanas, 110 posteriores al diagnóstico y 93 muestras previas al diagnóstico del conjunto de entrenamiento, y entrenamos un clasificador de regresión logística (LR) conjunto usando los valores de AMF para estas muestras; utilizamos un método establecido 54para este proceso con el fin de evitar el sobreajuste (Nota complementaria 1 ). Las muestras del conjunto de entrenamiento se dividieron aleatoriamente en dos grupos: uno para el ajuste del modelo y otro para la validación del modelo. El conjunto de ajuste del modelo se utilizó para entrenar un clasificador LR, y las puntuaciones del modelo se calcularon para el conjunto de validación del modelo. Este proceso se repitió para 1000 divisiones aleatorias diferentes del conjunto de entrenamiento, y se promediaron los puntajes del modelo en cada muestra para producir los resultados finales; Los resultados de cada división de muestra individual se muestran en los Datos complementarios 4 y la Fig. 3 complementaria . El clasificador final es, por lo tanto, un promedio de 1000 clasificadores LR construidos en diferentes divisiones del conjunto de entrenamiento. Este clasificador logró una sensibilidad del 88% para las muestras posteriores al diagnóstico y una sensibilidad del 91% para las muestras previas al diagnóstico con una especificidad elegida del 95% en el conjunto de entrenamiento (Tabla 1 , Datos complementarios 2 ). Después de la evaluación en el conjunto de entrenamiento, los parámetros del modelo se congelaron y no cambiaron para todos los análisis posteriores. Además, demostramos que los métodos alternativos de aprendizaje automático proporcionaban métricas de precisión similares (Nota complementaria 2 ), que el rendimiento del modelo seguía siendo idéntico incluso si se excluían las muestras posteriores al diagnóstico (Nota complementaria 3), el rendimiento del modelo se mantuvo alto incluso con un requisito mínimo de profundidad de lectura (Nota complementaria 5 ), con criterios de selección de marcadores más estrictos basados en el tejido canceroso (Nota complementaria 6 , ver más abajo).
Tabla 1 Precisión de PanSeer.
| Conjunto de entrenamiento | Equipo de prueba | ||||||
|---|---|---|---|---|---|---|---|
| Categoría | Total | # de muestras | Especificidad (%, IC 95%) | Sensibilidad (%, IC 95%) | # de muestras | Especificidad (%, IC 95%) | Sensibilidad (%, IC 95%) |
| Saludable | 414 | 207 | 94,7 (90,7–97,3) | 207 | 96,1 (92,5–98,3) | ||
| Post-diagnóstico | 223 | 110 | 88,2 (80,6–93,6) | 113 | 87,6 (80,1–93,1) | ||
| Pre-diagnóstico | 191 | 93 | 91,4 (83,8–96,2) | 98 | 94,9 (88,5–98,3) | ||
| 0-1 año antes del diagnóstico | 22 | 100 (84,6–100) | 21 | 95,2 (76,2–99,9) | |||
| 1 a 2 años antes del diagnóstico | 21 | 90,5 (69,6–98,8) | 23 | 95,7 (78,1–99,9) | |||
| 2 a 3 años antes del diagnóstico | 19 | 94,7 (74,0–99,9) | 31 | 93,6 (78,6–99,2) | |||
| 3 a 4 años antes del diagnóstico | 31 | 83,9 (66,3–94,6) | 23 | 95,7 (78,1–99,9) | |||
PanSeer detecta con precisión el cáncer en muestras posteriores al diagnóstico
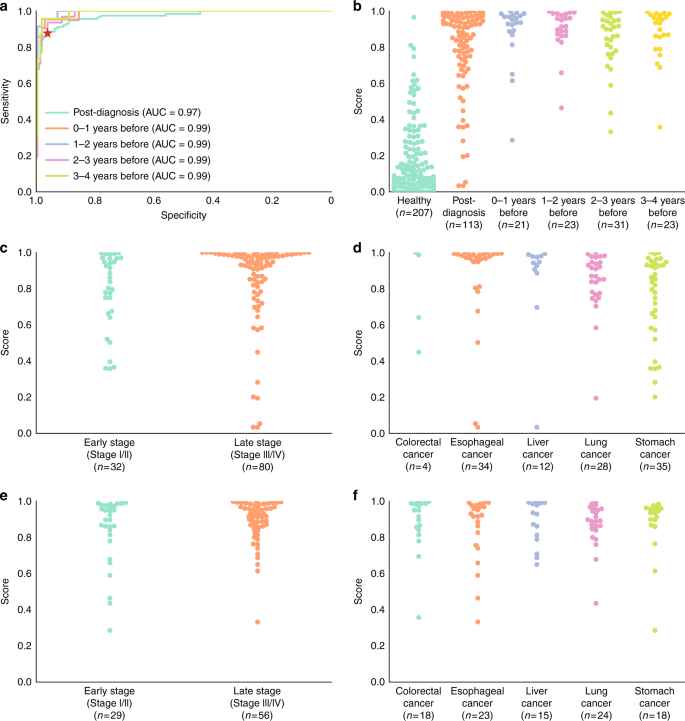
Aplicamos el clasificador LR a las 113 muestras posteriores al diagnóstico y 207 sanas en el conjunto de prueba de exclusión. La curva de características de funcionamiento del receptor (ROC) para el conjunto de prueba se muestra en la Fig. 2a . La sensibilidad general del clasificador fue del 88% en los pacientes con cáncer post diagnóstico (Tabla 1 , Fig. 2b , Fig. Suplementaria 6 ) con una especificidad del 96% (Tabla 1 , Fig. 2b , Fig. Suplementaria 6 ). La sensibilidad fue similar para las muestras de cáncer en estadio temprano y en estadio tardío (Fig. 2c , Suplementario Fig. 4 ), y varió del 75% en los cánceres colorrectales al 96% en los cánceres de pulmón (Fig. 2d, Figura complementaria 5 ). Si bien los falsos positivos probablemente representan clasificaciones erróneas por el ensayo PanSeer, es posible que algunos pacientes tengan cáncer no detectado que aún no se diagnosticó durante el estudio TZL; Tomamos un enfoque conservador y clasificamos estas muestras como falsos positivos. Detecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales
Todos los resultados presentados utilizaron solo las muestras del conjunto de prueba. Los puntos representan el puntaje de regresión logística (LR). a Curvas características del operador receptor (ROC) y valores de área bajo la curva (AUC) para PanSeer. La estrella roja muestra el valor de corte derivado del conjunto de entrenamiento. Se muestran curvas separadas para las muestras posteriores al diagnóstico y las muestras previas al diagnóstico (divididas por años antes del diagnóstico). b Puntuaciones LR para muestras de PanSeer por años antes del diagnóstico. c Puntuaciones LR para muestras de PanSeer por etapa de cáncer para muestras posteriores al diagnóstico. d Puntuaciones de LR para muestras de PanSeer por tejido de origen para muestras posteriores al diagnóstico. e Puntuaciones LR para muestras de PanSeer por etapa de cáncer en el momento del diagnóstico para muestras pre-diagnósticas. F Puntuaciones de LR para muestras de PanSeer por tejido de origen para muestras de pre-diagnóstico.
PanSeer detecta el cáncer hasta cuatro años antes del diagnóstico
Una característica importante de un ensayo de detección es diagnosticar el cáncer antes que los métodos convencionales 21 . Por lo tanto, evaluamos la capacidad del clasificador LR conjunto para detectar el cáncer en los 143 pacientes aparentemente sanos que luego fueron diagnosticados con cáncer dentro de los 4 años. En estos pacientes con cáncer previo al diagnóstico, observamos una sensibilidad general del 95% en el conjunto de pruebas de exclusión (tabla 1 , figura 2b , figura complementaria 6 ). La sensibilidad fue similar para los pacientes que finalmente fueron diagnosticados con cáncer en estadio temprano y en estadio tardío (Fig. 2e ), y osciló entre 91% en cáncer de esófago y 100% en cáncer de hígado (Fig. 2f) La sensibilidad parecía ser similar entre los pacientes diagnosticados de uno a cuatro años después, independientemente de la etapa del cáncer en el diagnóstico convencional (Figura 7 complementaria ).
Además, realizamos un análisis de covariables utilizando la prueba de Kruskal-Wallis (con pruebas post hoc) sobre la edad del paciente, el sexo del paciente, la fecha de recolección, el lugar de recolección, el estado de fumar, el estado de la enfermedad no cancerosa, el número de moléculas de ADN únicas observadas y el total la cantidad de ADN extraído para asegurar que otros factores clínicos o de recogida no estaban contribuyendo a la capacidad del ensayo de PanSeer para detectar el cáncer antes del diagnóstico convencional (complementario Figs. 8 – 18) Si bien se observó variabilidad entre diferentes subconjuntos de muestras, debido a la baja tasa de incidencia de cáncer, fue difícil establecer relaciones específicas entre las covariables y la precisión del ensayo. Después de las pruebas post-hoc, la única relación observada con un efecto en la precisión del ensayo fue una reducción en la especificidad para las muestras sanas recolectadas antes de 2010 (que representa aproximadamente una cuarta parte de las muestras sanas en este estudio), mientras que la sensibilidad se mantuvo constante (Fig. 10 suplementaria ); Es posible que el mayor tiempo de almacenamiento de estas muestras haya causado una especificidad reducida debido a la degradación del ADN (Figura complementaria 10) . ) A pesar de esta variabilidad, la sensibilidad general y la especificidad se mantuvieron altas independientemente del estado clínico de covariable; El ensayo PanSeer pudo utilizar señales de metilación para detectar cáncer hasta cuatro años antes del diagnóstico convencional, independientemente de las covariables clínicas.
Debido a la naturaleza retrospectiva de la TZL, no podíamos estar seguros de si las muestras de pre-diagnóstico clasificadas como normales se debían a una clasificación errónea por el ensayo PanSeer o si estos pacientes realmente estaban libres de cáncer al momento de la recolección de la muestra y si desarrollaron cáncer por completo después de la recolección de muestras de sangre; Tomamos un enfoque conservador y clasificamos estas muestras como falsos negativos.
Diferencias de metilación entre muestras de tejido y plasma.
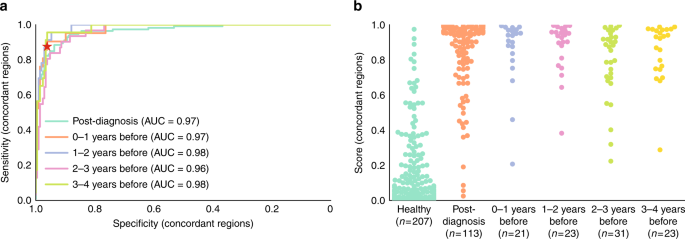
Si bien el ensayo PanSeer mostró una alta sensibilidad para la detección del cáncer tanto en muestras de plasma posdiagnóstico como prediagnóstico, observamos que algunos loci genómicos no mostraron cambios consistentes de metilación entre el tejido canceroso y las muestras de plasma canceroso. Estas regiones genómicas mostraron hipermetilación en tejido canceroso e hipometilación en plasma canceroso, o viceversa. Si bien esto probablemente se deba a diferencias intrínsecas en la metilación de tejidos y plasma, la variabilidad de los patrones de metilación del cáncer o la variación inherente en los patrones de metilación de muestreo en el ADN libre de células, existe la posibilidad de que estos patrones discordantes puedan indicar la presencia de un desconocido factor de confusión. 3 , Nota complementaria 6 ). Determinamos que 277 regiones genómicas mostraron metilación concordante entre tejido y plasma; cuando se usan solo estas 277 regiones genómicas para el modelado, la sensibilidad de ajuste de exclusión (85.0% para muestras posteriores al diagnóstico, 89.8% para muestras previas al diagnóstico) y la especificidad (95.1% para muestras sanas) se mantuvo alta (Fig. 3 , Nota complementaria 6 ) Si bien esto demuestra que el ensayo PanSeer puede detectar el cáncer en etapa temprana incluso con un conjunto objetivo más estrictamente elegido, se deben realizar más estudios longitudinales a gran escala para confirmar la detección temprana del cáncer en las muestras de pre-diagnóstico.
Todos los resultados presentados utilizaron solo las muestras del conjunto de prueba, y solo utilizaron regiones objetivo que mostraban hiper / hipometilación concordante entre las muestras de plasma de cáncer del conjunto de entrenamiento y las muestras de tejido canceroso. Los puntos representan el puntaje de regresión logística (LR). a Curvas características del operador receptor (ROC) y valores de área bajo la curva (AUC). La estrella roja muestra el valor de corte derivado del conjunto de entrenamiento. Se muestran curvas separadas para las muestras posteriores al diagnóstico y las muestras previas al diagnóstico (divididas por años antes del diagnóstico). b Puntuaciones LR por años antes del diagnóstico.
Discusión
En resumen, demostramos que se pueden detectar cinco tipos de cáncer mediante un análisis de sangre basado en la metilación del ADN hasta cuatro años antes del diagnóstico convencional. El ensayo PanSeer utiliza biomarcadores de metilación en toda su extensión al enfocarse de manera sensible en 10,613 sitios CpG en 477 regiones genómicas y utilizando un puntaje de conjunto aprendido por la máquina basado en cientos de regiones genómicas simultáneamente.
El ensayo PanSeer pudo detectar con éxito cinco tipos de cáncer utilizando un conjunto común de marcadores de metilación independientemente del tejido de origen. Como tal, los genes incluidos en el clasificador LR representan una firma epigenética central común a múltiples tipos de cáncer. Estos genes pueden merecer una mayor investigación en un contexto terapéutico, ya que un cambio en la regulación epigenética de estos genes parece ser un fenómeno de cáncer común. Si bien hemos demostrado la detección temprana del cáncer cuatro años antes del diagnóstico convencional mediante el uso de una cohorte longitudinal, nos gustaría enfatizar que el ensayo PanSeer probablemente no predice pacientes que luego desarrollarán cáncer. En cambio, lo más probable es que el ensayo identifique pacientes que ya tienen crecimientos cancerosos pero que permanecen asintomáticos a los métodos de detección actuales y al estándar de atención,21 . Detecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales
Detecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales
La posibilidad de una prueba de detección temprana de cáncer basada en la sangre se ha investigado recientemente a través de múltiples enfoques 30 , 55 , 56 , con un reciente consenso en torno a la alta utilidad de la metilación del ADN libre de células como marcador de cáncer 57 , 58 . Cuando se desarrolla una prueba de detección dirigida a una población de alto riesgo o de riesgo promedio, el costo es un factor crítico para garantizar la disponibilidad y adopción de la prueba. Si bien algunos estudios anteriores han demostrado que la metilación del ADN se puede utilizar para detectar y determinar de forma no invasiva el tejido de origen de un cáncer 45 , 59, estos estudios requirieron el uso de una gran cantidad de marcadores específicos de tejido y una gran cantidad de ADN de entrada, de modo que se requeriría más de un vial de sangre, lo que incurriría en un costo de prueba más alto. El ensayo PanSeer se desarrolló únicamente para detectar el cáncer, independientemente del tejido de origen, apuntando a un número limitado de regiones genómicas que comúnmente se metilan de manera aberrante a través de diferentes tipos de cáncer, lo que permite su uso como una posible pantalla de cáncer barata de primera línea; También requiere una cantidad relativamente pequeña de ADN de entrada (de un solo tubo de sangre). Por lo tanto, prevemos un contexto clínico en el que PanSeer podría usarse como una pantalla de primera línea; cualquier paciente que dé positivo en PanSeer se someterá a un examen de sangre reflejo más costoso y / o imágenes de seguimiento para permitir el mapeo de tejido de origen.
Se deben reconocer varias limitaciones de nuestro estudio. Primero, aunque este estudio fue longitudinal, el análisis fue retrospectivo e incluyó una proporción equivalente de cáncer y muestras sanas para permitir el desarrollo de un modelo de detección de cáncer preciso; aún no se ha establecido si el ensayo PanSeer mejoraría los resultados de los pacientes y requeriría un estudio prospectivo longitudinal. En segundo lugar, debido al marco temporal de TZL 27 , las técnicas modernas de preservación del plasma no se utilizaron durante la recolección de muestras, lo que condujo a varios grados de contaminación del ADN genómico y, en algunos casos, a una alta tasa de fracaso de la muestra; Además, solo 1 ml de plasma estaba disponible para cada muestra (Datos complementarios 2) Es probable que con las extracciones de sangre de 10 ml que se usan típicamente en los protocolos actuales y mejores técnicas de preservación del plasma, estén disponibles más moléculas de ADN de mayor calidad para el ensayo y la sensibilidad de detección pueda aumentar aún más. Tercero, el espectro de cánceres observado en la cohorte TZL (Fig. 1 ) no coincidía exactamente con la población general china 53; Estas diferencias podrían deberse a factores locales (como el estilo de vida, la contaminación o la composición genética). Cuarto, el número total de muestras de cáncer pre-diagnóstico en este estudio es limitado; Esta limitación es inevitable, ya que la tasa de incidencia de cáncer en una población sana es baja. Quinto, como se mencionó anteriormente, el ensayo PanSeer fue desarrollado únicamente para detectar cáncer independientemente del tejido de origen; Sería necesario utilizar un panel muy expandido que incorpore una gran cantidad de marcadores específicos de tejido para permitir el mapeo de tejido de origen. Sexto, debido a la naturaleza longitudinal de la TZL y al consentimiento otorgado por los pacientes, no pudimos obtener muestras de tejido de los pacientes pre-diagnósticos después de que fueron diagnosticados posteriormente; por lo tanto, para garantizar que las señales identificadas se derivan del tejido canceroso, Utilizamos 200 muestras primarias de tejido tumoral y de tejido normal de un proveedor comercial de muestras biológicas para elegir nuestros marcadores. Séptimo, debido a la naturaleza longitudinal y el diseño de la TZL, no pudimos obtener la información completa del estadio del cáncer para todos los pacientes pre-diagnósticos, ya que la base de datos de la TZL solo rastrea internamente si un paciente fue diagnosticado temprano (I / II) o etapa tardía (III / IV). Intentamos obtener información sobre la etapa para la mayor cantidad de pacientes posible, y hemos descrito por qué la información sobre la etapa no estaba disponible para cada paciente que faltaba estos datos en Datos complementarios ya que la base de datos TZL solo realiza un seguimiento interno si un paciente fue diagnosticado en una etapa temprana (I / II) o tardía (III / IV). Intentamos obtener información sobre la etapa para la mayor cantidad de pacientes posible, y hemos descrito por qué la información sobre la etapa no estaba disponible para cada paciente que faltaba estos datos en Datos complementarios ya que la base de datos TZL solo realiza un seguimiento interno si un paciente fue diagnosticado en una etapa temprana (I / II) o tardía (III / IV). Intentamos obtener información sobre la etapa para la mayor cantidad de pacientes posible, y hemos descrito por qué la información sobre la etapa no estaba disponible para cada paciente que faltaba estos datos en Datos complementarios 2 ; La razón más común para la falta de información sobre la etapa fue que el paciente murió de cáncer poco después del diagnóstico sin cirugía. Finalmente, como se discutió anteriormente, algunas regiones genómicas no mostraron patrones consistentes de metilación entre el tejido canceroso y el plasma canceroso; Si bien esto puede deberse a diferencias intrínsecas entre los tipos de muestra, es posible que exista algún factor de confusión desconocido. Llevamos a cabo análisis adicionales para eliminar la mayor incertidumbre posible (Nota complementaria 6 ), pero es necesaria una validación adicional de la detección del cáncer pre-diagnóstico en un estudio longitudinal grande para confirmar completamente estos resultados.
El ensayo PanSeer proporciona una demostración preliminar de la detección temprana de múltiples tipos de cáncer cuatro años antes del diagnóstico convencional de una manera sólida, y sienta las bases para un análisis de sangre no invasivo para la detección temprana del cáncer en un riesgo alto (o promedio) riesgo en el futuro) población. Si bien gran parte de la investigación actual sobre el cáncer se centra en el desarrollo de nuevas terapias, los estudios han demostrado que la detección temprana tiene el potencial de reducir tanto el costo del tratamiento como las tasas de mortalidad por cáncer en una cantidad significativa 21 . Los cinco tipos de cáncer estudiados aquí representan 261,530 muertes anuales por cáncer en los EE. UU. 4 y 2,1 millones de muertes anuales por cáncer en China 53; La detección temprana podría reducir en gran medida las muertes por estas enfermedades. Estudios recientes también han identificado que la detección temprana del cáncer podría reducir los costos de tratamiento del cáncer en $ 26 mil millones anuales (y también reducir la pérdida de productividad causada por el cáncer) 60 . En el futuro, para establecer completamente la utilidad clínica de PanSeer y validar completamente los resultados de la detección pre-diagnóstica de cáncer, esperamos proceder con un gran estudio prospectivo de individuos sanos para determinar si la detección de cáncer no invasivo puede reducir las muertes por cáncer en de manera rentable.
Métodos
Diseño del estudio Detecta el cáncer hasta cuatro años antes que los métodos convencionales Detecta el cáncer hasta cuatro años antes que los métodos convencionales
El Estudio Longitudinal de Taizhou (TZL) comenzó en julio de 2007, con el objetivo de reclutar a 200,000 personas en la ciudad de Taizhou, provincia de Jiangsu, China, y seguir a los participantes durante al menos 40 años 27 . Taizhou se encuentra en el centro de China, río abajo del río Yangtze. Si bien la población y la economía regionales son promedio para China, la incidencia de cánceres digestivos en Taizhou es alta, a niveles de 36.9, 29.8 y 30.0 por 100,000 personas-año para cáncer de esófago, gástrico y de hígado, respectivamente 61 . Según la vigilancia de las muertes por cáncer en 2010, la mortalidad por cáncer fue de 154.05 / 100,000 personas-año, casi el doble de la tasa de incidencia promedio para China 62 .
La encuesta de referencia del Estudio longitudinal de Taizhou tuvo lugar durante 2007–2016 en las áreas de Taixing, Gaogang y Hailing en Taizhou. Todos los hombres y mujeres de 30 a 75 años que vivían en estos distritos eran elegibles. Los ciudadanos potencialmente elegibles recibieron una carta de invitación de los líderes de la comunidad local y los trabajadores de la salud, con el apoyo del gobierno para extensas campañas publicitarias y promociones de salud. Se estableció un Centro de Coordinación Regional (RCC) y equipos de encuestas para la encuesta de línea de base, que consta de 40 miembros del personal a tiempo completo con calificaciones médicas y experiencia de trabajo de campo en los tres distritos estudiados. Todos los participantes fueron monitoreados indefinidamente en busca de cáncer a través de enlaces con registros locales de cáncer y bases de datos de seguros de salud. Los datos de exposición se recopilaron mediante cuestionarios de encuestas y mediciones físicas. Además, se recogieron muestras de sangre y otras muestras biológicas. Un total de 123,115 individuos han sido reclutados y el tiempo promedio de seguimiento es de 8.1 años.
Utilizando muestras del Estudio longitudinal de Taizhou (TZL), nos propusimos desarrollar un modelo de clasificación que pudiera identificar el cáncer de manera no invasiva antes de la aparición de síntomas de cáncer y el diagnóstico convencional de cáncer. El estudio fue aprobado por el Comité de Ética Humana de la Universidad de Fudan. Se obtuvo el consentimiento informado por escrito de todos los participantes del estudio antes de su inclusión en el TZL. Para muestras sanas, se requería que el individuo no fuera diagnosticado con cáncer durante el período de monitoreo (mínimo de 5 años). Para las muestras previas al diagnóstico, se requería que se determinara un diagnóstico positivo de cáncer de pulmón, hígado, estómago, esófago o colorrectal dentro de los 4 años posteriores a la extracción de sangre inicial. Para las muestras posteriores al diagnóstico, se requería un diagnóstico positivo de pulmón, hígado, estómago, esófago, o el cáncer colorrectal se determinó antes de la extracción de sangre inicial, y que los pacientes no habían recibido tratamiento previo. Los criterios de exclusión fueron información clínica incompleta, volumen plasmático inferior a 1 ml o presencia de hemólisis en plasma. Para todas las muestras, después del procesamiento y la secuenciación, se requería que se observaran al menos 200,000 moléculas de ADN mapeadas únicas en los datos de secuenciación, ya que cantidades menores indican una muestra de baja calidad.
El plan estadístico del estudio incorporó tamaños de grupo de 144 individuos de control y 144 pacientes de casos; este tamaño de muestra fue suficiente 63 , 64 para verificar que el ensayo PanSeer tenía una sensibilidad y especificidad esperadas del 75% con una potencia de 1 − β = 90% y un nivel de significancia de α = 0.05. Se incorporaron al estudio pacientes y casos adicionales de control debido a su disponibilidad.
Se recolectó un total de 1156 muestras de plasma de la cohorte TZL y los biobancos del hospital local de Taizhou para su inclusión en este estudio. En la cohorte TZL, un total de 575 participantes del estudio inicialmente sanos fueron diagnosticados posteriormente con cáncer colorrectal, esofágico, hepático, pulmonar o estomacal dentro de los 4 años. De estas 575 muestras previas al diagnóstico, se seleccionaron 191 muestras que pasaron los criterios de inclusión / exclusión del estudio, confirmaron el diagnóstico de cáncer mediante una revisión retrospectiva de los registros de hospitalización y la patología de la biopsia, y aprobaron métricas experimentales de control de calidad. 191 muestras sanas que superan los criterios de inclusión / exclusión del estudio y que coinciden con el momento de la recolección, el sexo, el grupo de edad de 10 años y el recuento único de moléculas de ADN mapeadas a las muestras de pre-diagnóstico se seleccionaron al azar de las 110, 1 ) Luego se recogieron 223 muestras de plasma posteriores al diagnóstico que superaron los criterios de inclusión / exclusión del estudio de los biobancos del hospital local de Taizhou; Luego se seleccionaron al azar 223 muestras sanas que pasaron los criterios de inclusión / exclusión del estudio y se combinaron por tiempo de recolección, sexo, grupo de edad de 10 años y recuento de moléculas de ADN mapeado único a las muestras posteriores al diagnóstico de las 110,501 muestras sanas TZL no diagnosticadas con cáncer por al menos 5 años.
Las muestras se dividieron aleatoriamente en un conjunto de entrenamiento sin autorización y un conjunto de prueba de exclusión para análisis de datos en una proporción de aproximadamente 50%: 50% (utilizando un generador de números aleatorios). Para el conjunto de entrenamiento, se seleccionaron 110 muestras de cáncer post diagnóstico, 93 muestras de pre diagnóstico y 207 muestras sanas (emparejadas por sexo, grupo de edad de 10 años, fecha de recolección y recuento de lectura mapeado único); Estas muestras se utilizaron para la construcción del modelo, y los parámetros del modelo se fijaron antes de cualquier análisis de las muestras del conjunto de prueba. Para el conjunto de pruebas de exclusión coincidente, se seleccionaron 113 muestras post diagnóstico, 98 muestras pre diagnóstico y 207 muestras sanas emparejadas. Para el conjunto de pruebas de exclusión, los resultados clínicos se ocultaron al clasificador hasta que se realizaron las llamadas.
Recolección de muestras de tejido.
Se adquirió ADN extraído de 160 muestras de tejido canceroso y 40 sanas de Biochain (D8235086-1, D8235090-1, D8235152-1, D8235248-1). El ADN se fragmentó usando el cizallamiento de Covaris hasta un tamaño medio de 150 pb para imitar el tamaño del ADN libre de células del plasma. Luego se reparó el ADN (New England Biolabs, E6050L).
Recolección de muestras de plasma.
Se recogieron muestras de sangre de 123,115 individuos de 2007 a 2014 y se separaron en plasma como parte del Estudio Taizhou Lontiduinal (TZL) 27 . Se extrajeron diez mililitros de sangre de cada paciente en un aspirador K2 EDTA y se almacenaron a 4 ° C hasta el final del día hábil. Las muestras de sangre se centrifugaron y el plasma se dividió en alícuotas en un criovial con código de barras para el almacenamiento a largo plazo a -80 ° C o menos. Se obtuvieron 578 muestras de plasma sanas y 221 muestras de plasma pre-diagnóstico del biobanco TZL en base a criterios de inclusión y exclusión. La edad del paciente al momento de la recolección de la muestra de sangre, el sexo del paciente, la fecha de diagnóstico del cáncer y el tejido de origen del cáncer se catalogaron para cada muestra (Datos complementarios 2) 357 muestras de plasma posteriores al diagnóstico recolectadas y almacenadas con el mismo protocolo y marco de tiempo que las muestras de TZL también se obtuvieron de biobancos del hospital local de Taizhou según los criterios de inclusión y exclusión, y se procesaron de la misma manera.
El ADN libre de células (ADNc) se extrajo de 1 ml de plasma utilizando el kit de ácido nucleico circulante QIAamp (Qiagen, 55114) y se eluyó en 50 μl de tampón de acuerdo con las instrucciones del fabricante, con la excepción de un período de incubación de 1 hora a 60 ° C durante El paso de lisis. Se utilizó ARN portador para mejorar la tasa de recuperación (según las sugerencias del fabricante). Las muestras se cuantificaron usando el método fluorométrico Qubit (usando 2 μL de volumen); nos gustaría señalar que es posible que el ARN portador pueda afectar el rendimiento de ADN observado, pero sesgaría todas las cuantificaciones por igual.
Ensayo PanSeer
El ADN del tejido y el cfDNA se convirtieron en bisulfito utilizando el kit de conversión de bisulfito de metilcódigo (ThermoFisher, MECOV50) de acuerdo con el protocolo del fabricante; Se usaron 30 μL de los 48 μL restantes para cada muestra, ya que este es el volumen máximo permitido por el kit Methylcode. Las muestras de ADN convertidas se procesaron luego en bibliotecas de secuenciación en Singlera Genomics y se secuenciaron en un Illumina NextSeq 500 en modo de 300 pb de extremo emparejado. Brevemente, el ADN convertido con bisulfito fue desfosforilado y ligado a un adaptador universal con un identificador molecular único (UMI). Después de una síntesis y purificación de la segunda cadena, el ADN se sometió a una PCR semi-dirigida a 595 regiones genómicas que cubren 11,787 sitios CpG. Después de una purificación, una segunda PCR agregó códigos de barras específicos de muestra y adaptadores de secuenciación Illumina de longitud completa.
El análisis de datos inicial se realizó como parte de la tubería de procesamiento de secuenciación de metilación Singlera estándar. Las lecturas se demultiplexaron utilizando el software Illumina bcl2fastq v2.20.0.422 ( https://support.illumina.com/sequencing/sequencing_software/bcl2fastq-conversion-software.html ). Para cada muestra, los archivos FASTQ de lectura de extremo emparejado se fusionaron en lecturas únicas usando PEAR v0.9.6 ( https://sco.h-its.org/exelixis/web/software/pear/doc.html ). Las lecturas en el archivo FASTQ fusionado se recortaron con el adaptador usando trim_galore v0.4.0 ( https://www.bioinformatics.babraham.ac.uk/projects/trim_galore/ ). La secuencia del identificador molecular único para cada lectura se agregó al nombre de la lectura usando UMI_tools v0.5.5 ( https://github.com/CGATOxford/UMI-tools) Las lecturas recortadas se alinearon luego con el genoma de referencia humano convertido en bisulfito (versión hg19) utilizando Bismark v0.17.0 ( https://www.bioinformatics.babraham.ac.uk/projects/bismark/ ) y Bowtie2 v2.3.1 ( http : //bowtie-bio.sourceforge.net/bowtie2/index.shtml ). Las muestras de plasma con menos de 200,000 moléculas de ADN mapeadas de forma exclusiva se excluyeron del procesamiento posterior debido a las bibliotecas de baja calidad; Si se extrajo una muestra, su muestra normal / cáncer también se eliminó para mantener un conjunto de muestras equilibrado. Esto dejó un conjunto de 191 muestras pre-diagnóstico, 223 muestras post-diagnóstico y 414 muestras sanas, así como 200 muestras de tejido sano y canceroso.
Para cada muestra restante, se asignaron lecturas alineadas a cada una de las 595 regiones objetivo cubiertas por el ensayo PanSeer en función de la posición genómica mapeada. La fracción de metilación promedio (AMF) se calculó para cada región objetivo sumando el número de citosinas observadas en todos los sitios CpG cubiertos y dividiendo por la profundidad de secuenciación total en todos los sitios CpG cubiertos en cada región según la fórmula:
donde I representa un sitio CpG en esta región objetivo, M es el número total de sitios CpG en esta región objetivo, N T, I representa el número de timinas observadas en el sitio CpG I , N C, I representa el número de citosinas observadas en Sitio de CpG i . Se proporciona un ejemplo gráfico de este cálculo en la figura complementaria 19 . Este proceso resultó en una matriz con 595 filas (una para cada región objetivo) y 1020 columnas (una para cada muestra); estos valores de AMF se han proporcionado como datos complementarios 5 .
Selección de marcador
Con el fin de garantizar que las regiones objetivo utilizadas para el análisis de ADN libre de células muestren metilación aberrante en el tejido canceroso, utilizamos el conjunto de 160 muestras de tejido canceroso y 40 muestras de tejido sano (incluidas 8 muestras de linfocitos primarios) obtenidas de Biochain. Para cada una de las 595 regiones objetivo, realizamos una prueba t con la corrección de pruebas múltiples Benjamini-Hochberg comparando los valores de AMF para muestras de tejido canceroso con los valores de AMF para muestras de tejido sano para cada tipo de tejido. 477 regiones genómicas que muestran valores de AMF estadísticamente significativamente diferentes (con p corregido-valor de corte α ≤ 0.05) en uno o más tipos de tejido fueron retenidos para el procesamiento aguas abajo; todas las demás posiciones se eliminaron de las matrices AMF. Estas 477 regiones genómicas resultantes correspondieron a 10,613 sitios CpG que podrían diferenciar las muestras de tejidos sanos y linfocitos sanos de Biochain de las muestras de tejido canceroso de Biochain (Figura complementaria 20 ). Para confirmar adicionalmente que los sitios elegidos realmente representaban una firma pan-cancerígena, verificamos que las regiones objetivo de PanSeer mostraban una metilación diferencial entre el tejido sano y el cáncer para otros tipos de tejidos en el conjunto de datos TCGA 28 disponible públicamente (figura complementaria 21 ).
Algoritmo de detección de cáncer
Para clasificar cada muestra de plasma como derivada de un paciente sano o paciente con cáncer, se construyó un clasificador de regresión logística (LR) utilizando las muestras del conjunto de entrenamiento; Para evitar el sobreajuste, se utilizó un enfoque de validación cruzada 54 . El procedimiento detallado utilizado para construir el clasificador se describe a continuación, y el código (con pseudocódigo en línea en los comentarios) se proporciona en la Nota complementaria 1 :
- 1)La matriz AMF fue el primer subconjunto para contener solo las columnas correspondientes a las muestras del conjunto de entrenamiento; Las muestras del conjunto de prueba de exclusión no se consideraron hasta que se finalizaron los parámetros del modelo.
- 2)Con el fin de garantizar que las regiones genómicas consideradas por el modelo estuvieran cubiertas en todas las muestras, cualquier región para la cual ninguna muestra no contuviera al menos una lectura se eliminó de la consideración en el modelo (es decir, cualquier fila de la matriz AMF con un valor faltante) fue eliminado de la matriz). Quedaron 471 de 477 regiones genómicas después de este paso de filtrado.
- 3)Se definió una matriz objetivo con una longitud igual al número de muestras de conjunto de entrenamiento. Si una muestra de conjunto de entrenamiento era correcta, su valor de matriz objetivo correspondiente se estableció en 0, mientras que si una muestra de conjunto de entrenamiento era cancerosa, su valor de matriz objetivo correspondiente se estableció en 1.
- 4)El conjunto de entrenamiento se dividió repetidamente al azar en una proporción del 50%: 50% en un conjunto de construcción de modelos y un conjunto de validación de modelos. Este proceso de división se repitió 1000 veces, lo que condujo a 1000 conjuntos diferentes de construcción y validación de modelos (todos los subconjuntos del conjunto de capacitación). Este procedimiento de validación cruzada permite la estimación de los parámetros del modelo usando solo la mitad del conjunto de entrenamiento y la validación de los parámetros del modelo usando la otra mitad del conjunto de entrenamiento.
- 5)Para cada conjunto de construcción de modelos, se utilizó el módulo LogisticRegression del paquete scikit-learn ( http://scikit-learn.org ) para los parámetros de aprendizaje automático para un clasificador LR que usa la matriz AMF como datos de entrenamiento y la matriz objetivo como objetivo valores. El solucionador liblinear implementado en scikit-learn ( http://www.csie.ntu.edu.tw/~cjlin/liblinear ) se utilizó para este proceso, y se utilizó una penalización LASSO para construir un modelo robusto; La función LogisticRegressionCV del paquete scikit-learn se utilizó para elegir el parámetro de penalización LASSO utilizando solo las muestras de construcción de modelos. Esto dio como resultado 1,000 ecuaciones LR diferentes (una para cada conjunto de construcción de modelos), que se usaron como un conjunto.
- 6)Estas ecuaciones fueron almacenadas. Cada ecuación tenía la forma:
PAGS= 1 –11 +mi– [ (∑nortei = 1METROyoXyo) +B]PAGS=1-11+mi-[(∑yo=1norteMETROyoXyo)+si](2)
donde P es la probabilidad de que una muestra dada sea cancerosa, n es el número de regiones genómicas cubiertas por el ensayo PanSeer, I es el haplotipo actual, X i es el valor de AMF de la i ésima región genómica, M i representa un coeficiente lineal ajustado por el módulo de aprendizaje automático LR, y B representa un coeficiente de intercepción ajustado por el módulo de aprendizaje automático LR.
- 1)Estas ecuaciones se utilizaron para calcular las puntuaciones de LR para cada conjunto de validación de modelo correspondiente; esto dio como resultado una matriz de puntajes LR, con cada muestra de conjunto de entrenamiento teniendo múltiples puntajes de todas las iteraciones donde era parte del conjunto de validación del modelo.
- 2)Se calculó un puntaje final de LR para cada muestra del conjunto de entrenamiento promediando todos los puntajes calculados cuando una muestra era parte del conjunto de validación del modelo. Se eligió un punto de corte de 0.583 basado en los puntajes finales del conjunto de entrenamiento para lograr una especificidad lo más cercana posible al 95%; Las muestras con puntuaciones superiores a este valor se consideraron cancerosas, mientras que las muestras con puntuaciones inferiores a este valor se consideraron sanas. La precisión del modelo se calculó para el conjunto de entrenamiento utilizando estos puntajes y puntos de corte promedio (Tabla 1 ). Este modelo de conjunto que consiste en el resultado promedio de 1000 ecuaciones se bloqueó antes de cualquier análisis del conjunto de prueba de exclusión.
- 3)Para todas las muestras de conjuntos de prueba de exclusión, las puntuaciones de LR se calcularon utilizando las 1000 ecuaciones, y se calculó una puntuación final promediando estas puntuaciones de LR individuales. El puntaje final se comparó con el límite del conjunto de entrenamiento, y se calculó la precisión del modelo para el conjunto de prueba de exclusión utilizando estos puntajes promedio (Tabla 1 ).
Al utilizar este enfoque clasificador de conjunto, el riesgo de sobreajuste se reduce en gran medida (ya que se puede asegurar que el rendimiento del conjunto de entrenamiento y del conjunto de prueba sea similar). Las ecuaciones completas para el clasificador de regresión logística de conjunto (incluidos los coeficientes, las intersecciones y los puntos de corte) se presentan en los Datos complementarios 3 , y el código fuente se ha incluido en la Nota complementaria 1 . Además, este mismo procedimiento se repitió utilizando un clasificador de Análisis discriminante lineal en lugar de Regresión logística para demostrar que los resultados son independientes del método de aprendizaje automático elegido; El código fuente y los resultados se presentan en la Nota complementaria 2. Para demostrar aún más la robustez de este enfoque, también determinamos que la puntuación de la regresión logística no se vio afectada ni por el número de valores faltantes en una muestra ni por su tasa de conversión de bisulfito (Figuras suplementarias 22 , 23 ). También repetimos el modelo de regresión logística con dos restricciones adicionales para confirmar que las señales de metilación observadas en pacientes con diagnóstico previo y posterior al diagnóstico derivaron de cáncer; incluso con un requisito mínimo de profundidad de lectura (Nota complementaria 5 ) o utilizando criterios de selección de marcadores más estrictos basados en el tejido canceroso (Nota complementaria 6 ), el rendimiento del modelo se mantuvo alto.
análisis estadístico
Se utilizaron medias y desviaciones estándar o medianas y rangos para resumir variables continuas, mientras que números enteros y porcentajes se utilizaron para resumir variables categóricas. Las métricas de precisión se calcularon para cada conjunto de muestras y subconjunto en función de las covariables de muestra. La sensibilidad se define como verdaderos positivos / (verdaderos positivos + falsos negativos). La especificidad se define como verdaderos negativos / (verdaderos negativos + falsos positivos). Los intervalos de confianza binomiales para la sensibilidad y especificidad se calcularon utilizando el método Clopper-Pearson. Para determinar si alguna muestra de covariables afectaba el rendimiento del ensayo, se utilizó la prueba Kruskal-Wallis H para comparar los puntajes del modelo para cada categoría (saludable, pre-diagnóstico y post-diagnóstico) en cada covariable analizada (Figuras complementarias 8 –18 ); si es necesario, se utilizó la prueba U de Mann-Whitney para realizar pruebas post-hoc.
Límite de estudio de detección
Para medir el límite analítico de detección del ensayo PanSeer, se construyó un conjunto de muestras espigadas que consisten en una mezcla de ADN de línea celular de cáncer y plasma sano; se determinó si cada nivel de inserción se podía separar del plasma basal sano. Para crear las muestras espigadas, se adquirió ADN de la línea celular HT-29 de ATCC y se cortó a aproximadamente 150 pb (Covaris). El ADN luego se purificó y se concentró usando perlas Ampure. El plasma de múltiples individuos sanos se combinó para usar como referencia en el estudio de límite de detección. HT-29 cizallado se introdujo en el plasma agrupado en relaciones molares del 0% (línea de base), 0.1, 0.5, 1.0, 5.0 y 10%, con seis réplicas técnicas para cada relación de inserción.
El ensayo PanSeer se ejecutó luego en las muestras de espiga. Para evaluar el límite de detección analíticamente, debido a la variación en los niveles de metilación en todo el genoma, se eligieron cuatro muestras de referencia como muestras de entrenamiento para determinar el nivel de metilación de fondo observable en plasma sano en cada región genómica. Para cada región genómica individual, se determinó un valor de corte utilizando estas cuatro muestras de entrenamiento de referencia; Este límite se estableció en tres desviaciones estándar por encima del valor medio de metilación observado en las muestras de referencia. Para las dos muestras restantes de inserción de 0% y todas las muestras de adición de 0.1–10%, el número de regiones genómicas para las cuales cada muestra estaba por encima del valor de corte calculado se totalizó y se representó en la figura complementaria 1 . ; todas las réplicas para todas las muestras de espiga se podían distinguir de las muestras de línea de base, con relaciones molares de espiga más altas que resultaban en más regiones genómicas por encima del valor de línea de base. El código detallado para el análisis del límite de detección se proporciona en la Nota complementaria 4.
Resumen de informes
Más información sobre el diseño de la investigación está disponible en el Resumen de Informes de Investigación de la Naturaleza vinculado a este artículo.
Disponibilidad de datos
Los datos de este estudio, incluidas las matrices de metilación, están disponibles en el texto principal, materiales complementarios, conjuntos de datos complementarios, o se han depositado en GitHub en el repositorio NCOMMS-20-10056-T [ https://github.com/ncomms- 20-10056-t / ncomms-20-10056-t ]. Los datos completos de secuenciación genética no se incluyeron en el consentimiento informado, por lo tanto, solo se ha publicado el estado de metilación en cada posición genómica. El conjunto de datos TCGA está disponible en el Portal de datos de GDC [ https://portal.gdc.cancer.gov/ ].
Disponibilidad de código
El código Python utilizado en este estudio se ha depositado en GitHub en el repositorio NCOMMS-20-10056-T [ https://github.com/ncomms-20-10056-t/ncomms-20-10056-t ].