La IA también predice con precisión las estructuras de ARN

La inteligencia artificial también predice con precisión las estructuras de ARN
Publicado en por el Dr. Francis Collins
Los investigadores demostraron recientemente que una computadora podría “aprender” de muchos ejemplos de plegamiento de proteínas para predecir la estructura 3D de las proteínas con gran velocidad y precisión. Ahora, un estudio reciente de la revista Science muestra que una computadora también puede predecir las formas 3D de las moléculas de ARN [1]. Esto incluye el ARNm que codifica proteínas y el ARN no codificante que realiza una variedad de funciones celulares.
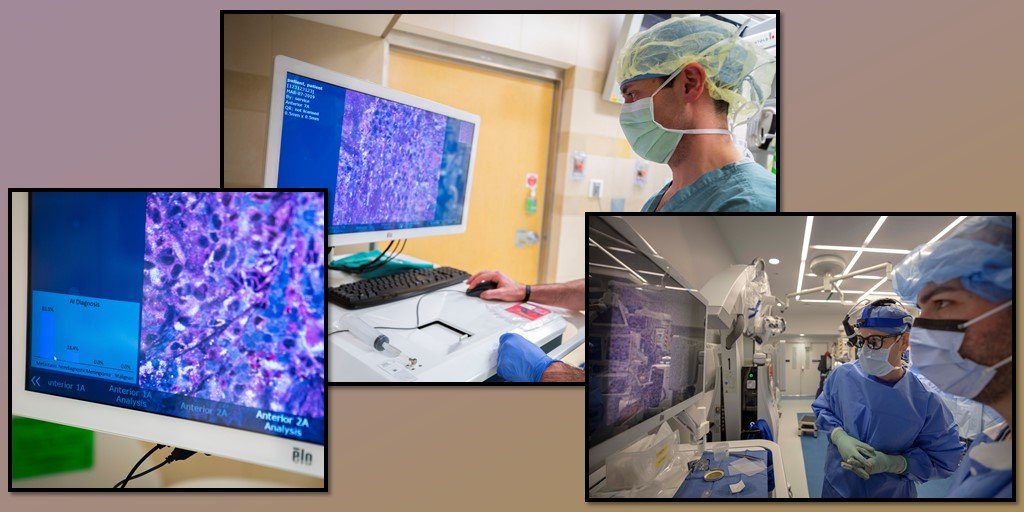
La inteligencia artificial acelera el diagnóstico de tumores cerebrales. Arriba, el médico revisa la muestra de tumor digitalizada en el quirófano; a la izquierda, el programa de IA predice el diagnóstico; a la derecha, los cirujanos revisan los resultados casi en tiempo real.
Crédito: Joe Hallisy, Medicina de Michigan, Ann Arbor
Este trabajo marca un importante avance científico básico. Las terapias de ARN, desde las vacunas COVID-19 hasta los medicamentos contra el cáncer, ya han beneficiado a millones de personas y ayudarán a muchas más en el futuro. Ahora, la capacidad de predecir formas de ARN de forma rápida y precisa en una computadora ayudará a acelerar la comprensión de estas moléculas críticas y ampliar sus usos en la atención médica.
Al igual que las proteínas, las formas de las moléculas de ARN monocatenario son importantes por su capacidad para funcionar correctamente dentro de las células. Sin embargo, se sabe mucho menos sobre estas estructuras de ARN y las reglas que determinan sus formas precisas. Los elementos de ARN (bases) pueden formar pares internos con enlaces de hidrógeno, pero el número de posibles combinaciones de emparejamientos es casi astronómico para cualquier molécula de ARN con más de unas pocas docenas de bases.
Con la esperanza de hacer avanzar el campo, un equipo dirigido por Stephan Eismann y Raphael Townshend en el laboratorio de Ron Dror, Universidad de Stanford, Palo Alto, CA, buscó un enfoque de aprendizaje automático conocido como aprendizaje profundo. Está inspirado en cómo las redes neuronales de nuestro propio cerebro procesan la información, aprendiendo a enfocarse en algunos detalles pero no en otros.
En el aprendizaje profundo, las computadoras buscan pat
rones en los datos. A medida que comienzan a “ver” relaciones complejas, algunas conexiones en la red se fortalecen mientras que otras se debilitan.

Arriba: las moléculas de proteína están formadas por aminoácidos unidos como cuentas en una cuerda. Abajo: para volverse activas, las proteínas deben torcerse y doblarse en su configuración final o “nativa”. Crédito: NIGMS.
Una de las cosas que hace que el aprendizaje profundo sea tan poderoso es que no se basa en nociones preconcebidas. También puede detectar características y patrones importantes que los humanos no pueden detectar. Pero, a pesar de lo exitoso que ha sido este enfoque para resolver muchos tipos diferentes de problemas, se ha aplicado principalmente a áreas de la biología, como el plegamiento de proteínas, en las que se disponía de una gran cantidad de datos para que los investigadores pudieran entrenar las computadoras.
Ese no es el caso de las moléculas de ARN. Para solucionar este problema, el equipo de Dror diseñó una red neuronal a la que llaman ARES. (No, no es el dios griego de la guerra. Es la abreviatura de Atomic Rotationally Equivariant Scorer).
Para empezar, los investigadores entrenaron a ARES con solo 18 pequeñas moléculas de ARN cuyas estructuras se habían determinado experimentalmente. Le dieron a ARES estos modelos estructurales especificados solo por su estructura atómica y elementos químicos.
La siguiente prueba fue para ver si ARES podía determinar a partir de este pequeño conjunto de entrenamiento el mejor modelo estructural para secuencias de ARN que nunca antes había visto. Los investigadores lo pusieron a prueba con moléculas de ARN cuyas estructuras se habían determinado más recientemente.
ARES, sin embargo, no crea las estructuras en sí. En cambio, los investigadores le dan a ARES una secuencia y al menos 1.500 posibles estructuras 3D que podría tomar, todas generadas con otro programa de computadora. Según los patrones del conjunto de entrenamiento, ARES puntúa cada una de las estructuras posibles para encontrar la que predice que está más cerca de la estructura real. Sorprendentemente, lo hace sin que se le proporcione información previa sobre características importantes para determinar las formas del ARN, como nucleótidos, restricciones estéricas y enlaces de hidrógeno.
Resulta que ARES supera constantemente a los humanos y a todos los demás métodos anteriores para producir los mejores resultados. De hecho, superó al menos otros nueve métodos para llegar a la cima en un concurso de rompecabezas de ARN en toda la comunidad. También puede hacer predicciones sobre moléculas de ARN que son significativamente más grandes y más complejas que aquellas sobre las que se entrenó.
El éxito de ARES y este enfoque de aprendizaje profundo ayudarán a dilucidar las moléculas de ARN con implicaciones potencialmente importantes para la salud y la enfermedad. Es otro ejemplo convincente de cómo el aprendizaje profundo promete resolver muchos otros problemas en biología estructural, química y ciencias de los materiales cuando, al principio, se sabe muy poco.
Referencia :
[1] Aprendizaje profundo geométrico de la estructura del ARN . Townshend RJL, Eismann S, Watkins AM, Rangan R, Karelina M, Das R, Dror RO. Ciencias. 27 de agosto de 2021; 373 (6558): 1047-1051.
Vínculos :
Biología estructural (Instituto Nacional de Ciencias Médicas Generales / NIH)
Las estructuras de la vida (Instituto Nacional de Ciencias Médicas Generales / NIH)
Biología del ARN (NIH)
Laboratorio de Dror (Universidad de Stanford, Palo Alto, CA)
Apoyo de los NIH: Instituto Nacional del Cáncer; Instituto Nacional de Ciencias Médicas Generales


