ADN en (aún más) detalle
ADN en (aún más) detalle, las nuevas tecnologías aceleran las imágenes de alta resolución de los cromosomas humanos en células individuales
Las estructuras intrincadas y fuertemente enrolladas de los cromosomas condensan alrededor de dos metros de ADN, por lo que el manual de instrucciones completo para el crecimiento de un humano se puede meter dentro de un núcleo celular de solo 10 micras de ancho.
“Esto es como colocar algo de la altura del Empire State Building en un espacio no más ancho que un fideo de espagueti”, dijo Huy Quoc Nguyen , investigador en genética de la Facultad de Medicina de Harvard.
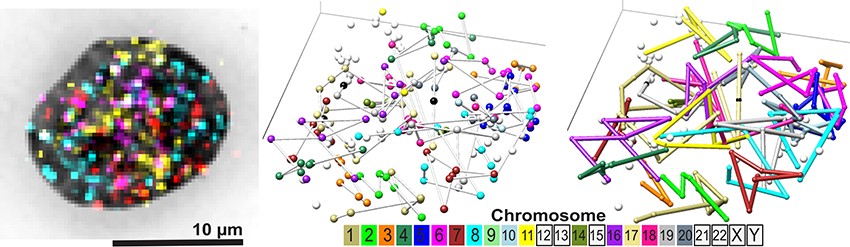
OligoFISSEQ ilumina más de 100 regiones genómicas en núcleos de células humanas. Imagen: laboratorio de Wu
Esas estructuras pueden afectar el comportamiento del ADN. Por ejemplo, los cambios dinámicos en la forma en que un cromosoma se retuerce y pliega pueden determinar cuánto se activa o desactiva un gen individual en una determinada célula, tejido, órgano o persona. ADN en (aún más) detalle
Exactamente cómo funciona este proceso, y cómo los defectos estructurales y las variaciones pueden conducir a problemas de salud, desde defectos de nacimiento hasta cáncer, sigue siendo turbio.
Obtenga más noticias de HMS aquí
Para comprender mejor cómo funciona la estructura del genoma, los científicos han estado desarrollando tecnologías de imágenes que pueden tomar instantáneas y videoclips de grandes franjas de cromosomas en las células a resoluciones cada vez más altas.
El objetivo final es observar genomas en su totalidad a escalas de solo unos pocos nanómetros.
Ahora, un equipo dirigido por genetistas en el Instituto Blavatnik en HMS, el Instituto Wyss de Ingeniería Biológicamente Inspirada y el Instituto de Ciencia y Tecnología de Barcelona (BIST) ha dado un paso más en esta odisea visual.
Los investigadores describen en Nature Methods un conjunto de técnicas denominadas colectivamente OligoFISSEQ (suena como “físico”).
OligoFISSEQ combina Oligopaints , sondas diseñadas por computadora que unen etiquetas fluorescentes a secuencias de ADN específicas, desarrolladas en el laboratorio de Ting Wu en HMS y Wyss, y secuenciación fluorescente in situ, o FISSEQ , un método desarrollado en Wyss que revela las ubicaciones de miles de moléculas de ARN en una célula dada, así como sus secuencias genéticas.
El resultado: los investigadores ahora pueden visualizar rápidamente más regiones del genoma a la vez en células humanas y crear mapas 3D de la organización cromosómica que son más completos y detallados que antes.
ADN en (aún más) detalle
Hasta ahora, los métodos de imágenes del genoma se basaban en la hibridación, que capturaba, a lo sumo, un puñado de objetivos a la vez en las células que no se dividían, a menudo centrándose en uno o dos cromosomas.
“Ahora conocemos muchas más ‘direcciones’ donde residen regiones específicas de ADN en tres dimensiones dentro del núcleo”, dijo Nguyen, quien es coautor del artículo con Shyamtanu Chattoraj del laboratorio Wu y David Castillo de BIST. ADN en (aún más) detalle
Como prueba de principio, el equipo del estudio utilizó OligoFISSEQ para apuntar hasta 249 regiones genómicas distribuidas en todos los cromosomas en cientos de células derivadas de la piel humana. El equipo también mapeó 46 regiones del cromosoma X con mayor detalle.
Los investigadores mapearon las ubicaciones de los objetivos genéticos a lo largo de cada cromosoma en tres dimensiones. Imágenes: Wu lab
Los investigadores diseñaron OligoFISSEQ para que sea compatible con muchas otras tecnologías comunes para investigar la biología y la bioquímica.
En el documento, describen cómo la combinación de OligoFISSEQ con OligoSTORM , otra técnica del laboratorio de Wu, aumenta la velocidad con la que los científicos pueden visualizar múltiples regiones de cromosomas grandes a una súper resolución. Por el momento, esas regiones pueden variar en longitud de decenas de miles a millones de bases, las “letras” del código de ADN.
“Esperamos que OligoFISSEQ nos acerque un paso más a la obtención de imágenes de genomas humanos completos con resoluciones limitadas por difracción y súper resoluciones”, dijo Wu, profesor de genética en HMS y miembro asociado de la facultad en Wyss.
“Darle a OligoFISSEQ la capacidad de combinarse con otras tecnologías debería extender aún más su utilidad”, dijo.
ADN en 3D
“En pocas palabras, OligoFISSEQ es como su mercado de compras único para la visualización del genoma”, coincidió Chattoraj. “Tendrás todos los ingredientes y podrás cocinar tu comida de la manera que quieras”.
Wu es coautor principal del artículo con Marc Marti-Renom en BIST y la Institución Catalana de Investigación y Estudios Avanzados (ICREA) en Barcelona.
La idea detrás de OligoFISSEQ se originó con los coautores Son Nguyen y Evan Daugharthy del laboratorio Wu. Nguyen ahora está en la Universidad de Pennsylvania, mientras que Daugharthy trabaja en ReadCoor, Inc., una compañía basada en FISSEQ.
La Universidad de Harvard ha presentado solicitudes de patentes relacionadas con Oligopaints y métodos relacionados basados en oligo para obtener imágenes del genoma. Wu y Daugharthy tienen participaciones en ReadCoor. Wu también tiene una colaboración de investigación con Bruker Nano, Inc. Los posibles conflictos de intereses para el coautor George Church, el profesor Robert Winthrop de genética en HMS y un miembro fundador de la facultad del Wyss, se enumeran aquí